Medical AI 勉強会 Part2 "ECG解析+転移学習"
論文を基に1-D CNN・転移学習・ECGデータ処理法を実装法を中心に学ぶ。
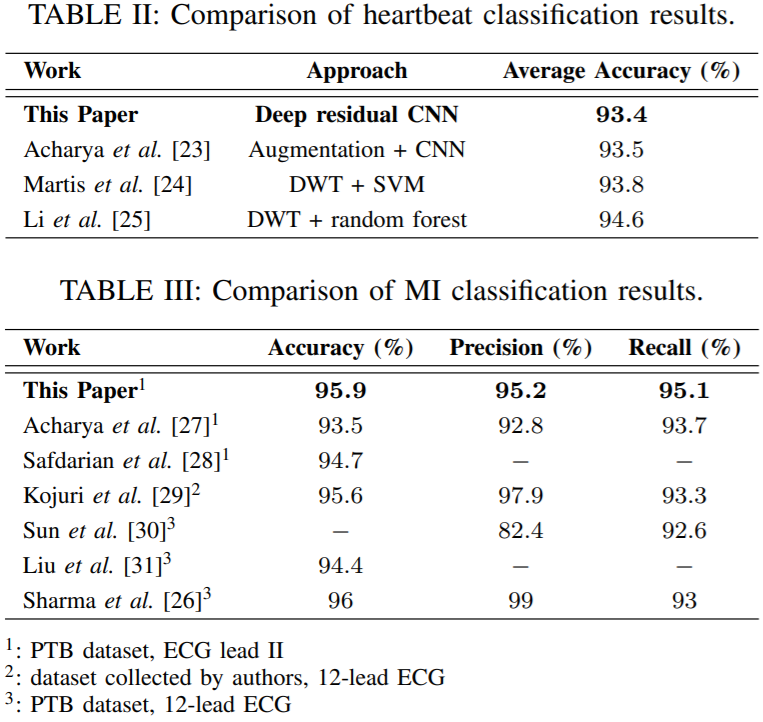
Abstract
ECG Heartbeat Classification: A Deep Transferable Representation
従来のECG解析に関する機械学習メソッドは異なるタスクごとに独立したものであったが、この論文ではタスクごとに知識を再利用できないかを検証することを目的としている。
まずMIT-BIHデータセットで不整脈分類タスク(5クラス)について学習させる。その後得られた学習済みNN(転移学習)に対してPTBデータセットで心筋梗塞2値分類タスクについて学習させた。
結果、平均Accuracyは不整脈分類タスクでは 93.4% で心筋梗塞2値分類タスクでは 95.9%と高精度な予測器を生成することができた。この結果から不整脈分類タスクの知識をうまく心筋梗塞2値分類タスクに転移させたことができたと主張している。
以下のような手順でECG波形の前処理を行ってます
- ECGデータを10秒間ごとのWindowに分けてそのうちの一つのWindowをとってくる
- 0~1に正規化する
- 極大値をすべて見つける
- 0.9以上の極大値をR-peakとする
- RR間隔の平均値を求め、その間隔をWindow長のTとする
- それぞれのR-peakから1.2Tだけデータをとる
- あらかじめ指定したデータ長に満たないデータを0で埋める(Zero-padding)
これら前処理済みデータセットはすでにKaggleに公開されています。
https://www.kaggle.com/shayanfazeli/heartbeat
実際のデータに学習モデルを適応するためには入力データの形式を学習時のそれと合わせる必要があります。実際に得られるECGデータはサンプリング周波数も異なるので上の前処理に加えてそこも調節する必要があります。
学習データセットは125Hzなのでまずはその周波数に従ってリサンプリングしましょう。
チュートリアル用に360HzでサンプリングされたScipyのECGデータを使います。
import numpy as np
from scipy.misc import electrocardiogram
import matplotlib.pyplot as plt
V = electrocardiogram()
Hz = 360 # 360Hzだから
T = np.arange(ecg.size) * 1000 / Hz
plt.figure(figsize=(10,7))
plt.plot(T, V)
plt.xlabel("[ms]")
plt.ylabel("[mV]")
plt.xlim(0, 10000)
plt.ylim(-1.1, 2.0)
plt.show()
まずは125Hzでリサンプリングします。
from scipy import interpolate
def resample(T, V, Hz=125, kind='linear'):
f = interpolate.interp1d(T,V,kind=kind)
T = np.arange(np.min(T), np.max(T), 1000/Hz)
V = f(T)
return T, V
T_new, V_new = resample(T, V)
plt.figure(figsize=(10,7))
plt.plot(T, V)
plt.plot(T_new, V_new)
plt.xlabel("[ms]")
plt.ylabel("[mV]")
plt.xlim(0, 10000)
plt.ylim(-1.1, 2.0)
plt.show()
ほぼ一致していることが分かります。それでは1.からみていきましょう。
- ECGデータを10秒間ごとのWindowに分けてそのうちの一つのWindowをとってくる
def split(T, V, window=10):
Hz = int(1000 / (T[1] - T[0]))
Ts = []
Vs = []
for i in range(0, len(T), window*Hz):
if T[i + window * Hz - 1:i + window * Hz]:
Ts.append(T[i:i+window*Hz])
Vs.append(V[i:i+window*Hz])
else:
Ts.append(T[i:])
Vs.append(V[i:])
return Ts, Vs
Ts, Vs = split(T_new, V_new)
plt.figure(figsize=(10,7))
for T, V in zip(Ts,Vs):
plt.plot(T, V)
plt.xlabel("[ms]")
plt.ylabel("[mV]")
plt.show()
- 0~1に正規化する
def normalize(V):
return (V-np.min(V))/(np.max(V)-np.min(V))
T_new, V_new = Ts[0], Vs[0]
V_new = normalize(V_new)
plt.figure(figsize=(10,7))
plt.plot(T_new, V_new)
plt.xlabel("[ms]")
plt.ylabel("[mV]")
plt.show()
- 極大値をすべて見つける
- 0.9以上の極大値をR-peakとする
from scipy.signal import find_peaks
def find_R_peaks(V, threshold=0.9):
R_peaks, _ = find_peaks(V, height=threshold)
return R_peaks
R_peaks = find_R_peaks(V_new)
plt.figure(figsize=(10,7))
plt.plot(T_new, V_new)
plt.scatter(T_new[R_peaks], V_new[R_peaks], color='r')
plt.xlabel("[ms]")
plt.ylabel("[mV]")
plt.show()
このデータではThreshold=0.9というのはあまりよくないようですが、論文の通りにいきましょう。
- RR間隔の平均値を求め、その間隔をWindow長のTとする
def find_median_interval(R_peaks):
return np.mean(np.diff(R_peaks)) # index
interval = find_median_interval(R_peaks)
- それぞれのR-peakから1.2Tだけデータをとる
- あらかじめ指定したデータ長に満たないデータを0で埋める(Zero-padding)
def extract_beats(T, V, R_peaks, interval, max_duration=187):
window = int(1.2*interval) # index
beats = []
durations = []
for peak in R_peaks:
beat = np.zeros(max_duration) # 固定長の空の行列をつくっとく
if peak + window <= len(V): # R_peakからWindow長データを取り切れる前提をおく
if window > max_duration: # Window長が指定した固定長を超えている場合
duration = [T[peak],T[peak+max_duration-1]]
beat = V[peak:peak+max_duration]
beats.append(beat)
durations.append(duration)
else:
duration = [T[peak],T[peak+window-1]]
beat[:window] = V[peak:peak+window]
beats.append(beat)
durations.append(duration)
return np.array(beats), durations # 抽出された心拍データとその始まりと終わりの時間を返す
beats, durations = extract_beats(T_new, V_new, R_peaks, interval)
print("Shape of the extracted beats data: ", beats.shape)
def ecg_with_beats(T,V,durations):
fig = plt.figure(figsize = (10,7))
ax = fig.add_subplot(111)
for i in range(len(durations)):
duration = durations[i]
ax.axvspan(duration[0], duration[1],color="coral" if i%2 == 0 else "lime" ,alpha=0.3)
ax.plot(T,V)
plt.xlabel("[ms]")
plt.ylabel("[mV]")
plt.show()
return
ecg_with_beats(T_new, V_new, durations)
plt.figure(figsize=(10,7))
for beat in beats:
plt.plot(beat)
plt.xlabel("index")
plt.ylabel("[mV]")
plt.show()
上手く心拍を抽出できていることがわかります。
最後に以上の処理をpreprocess関数にまとめてみましょう。
def preprocess(T, V, Hz=125, max_duration=187):
T, V = resample(T, V, Hz)
Ts, Vs = split(T, V)
Beats = []
Durations = []
for T, V in zip(Ts, Vs):
V = normalize(V)
R_peaks = find_R_peaks(V)
if len(R_peaks) >= 2:
interval = find_median_interval(R_peaks)
beats, durations = extract_beats(T, V, R_peaks, interval)
if len(beats) >= 1:
Beats.append(beats)
Durations += durations
Beats = np.vstack(Beats)
return Beats, Durations
beats, durations = preprocess(T, V)
print("Shape of the extracted beats data: ", beats.shape)
ecg_with_beats(T, V, durations)
ほんの一部の心拍が抽出されていることが分かります。この論文大丈夫か心配になってきましたね。
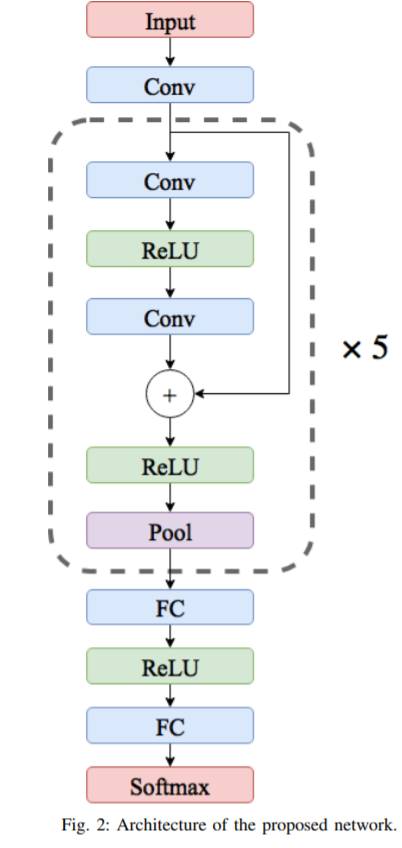
https://github.com/CVxTz/ECG_Heartbeat_Classification
論文にのってるモデルを少し変えたやつ(Residual blockなしバージョン)
1D-Convolution layer
"all convolution layers are applying 1-D convolution through time and each have 32 kernels of size 5"
- カーネル:入力にかける行列のこと、今回は1次元。32 Kernalsはカーネルの層数を意味するのでKerasだったら
filters = 32にあたる。 - サイズ:カーネルのWindow長。今回は一次元。size 5はKerasだったら
kernel_size = 5にあたる。
二次元畳み込み層よりもパラメータ数はもちろん少ない。今回は入力が一次元なので一次元畳み込み層で自然。
畳み込み層ついて詳しくは https://towardsdatascience.com/types-of-convolution-kernels-simplified-f040cb307c37
Dataset
https://www.kaggle.com/shayanfazeli/heartbeat のデータセットを使います。前処理済み最高長187の心拍がCSVファイルで格納されています。188番目のカラムにはその心拍のラベル(心室期外収縮や心筋梗塞など)が整数クラスで入ってます。
MITBIHのAnnotationは以下のようになってます。

N,S,V,F,Qはそれぞれ0,1,2,3,4クラスに対応しています。実際にデータセットを見てみましょう。
import pandas as pd
df_train = pd.read_csv("/content/drive/My Drive/kaggle_ECG/mitbih_train.csv", header=None) # 自分のGoogle driveにでもデータセットダウンロード
print("Data shape: ", df_train.shape)
print("All classes (shown in 188th column): ", df_train.iloc[:,187].unique())
plt.figure(figsize=(10,7))
for beat in df_train.iloc[:5,:].values:
plt.plot(beat)
plt.xlabel("index")
plt.ylabel("[mV]")
plt.show()
論文記載のアルゴリズムに従って前処理されていることが分かります。
MITBIHデータセットでまずは学習します。github借りパくです。
from keras import optimizers, losses, activations, models
from keras.callbacks import ModelCheckpoint, EarlyStopping, LearningRateScheduler, ReduceLROnPlateau
from keras.layers import Dense, Input, Dropout, Convolution1D, MaxPool1D, GlobalMaxPool1D, GlobalAveragePooling1D, \
concatenate
from sklearn.metrics import f1_score, accuracy_score
df_train = pd.read_csv("/content/drive/My Drive/kaggle_ECG/mitbih_train.csv", header=None)
df_train = df_train.sample(frac=1)
df_test = pd.read_csv("/content/drive/My Drive/kaggle_ECG/mitbih_test.csv", header=None)
Y = np.array(df_train[187].values).astype(np.int8)
X = np.array(df_train[list(range(187))].values)[..., np.newaxis]
Y_test = np.array(df_test[187].values).astype(np.int8)
X_test = np.array(df_test[list(range(187))].values)[..., np.newaxis]
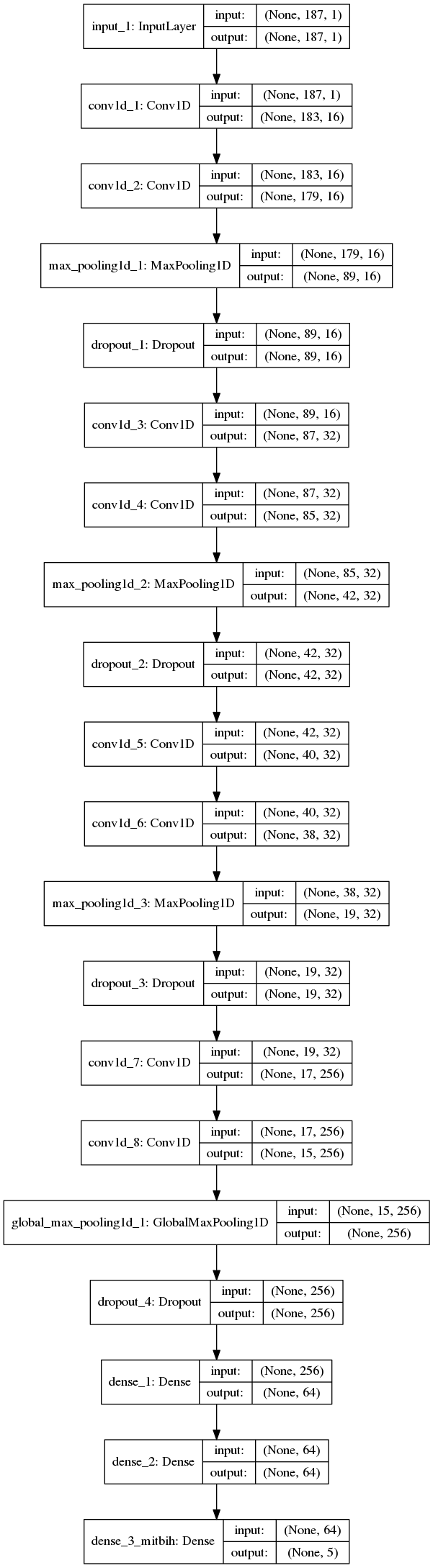
def get_model_mitbih():
nclass = 5
inp = Input(shape=(187, 1))
img_1 = Convolution1D(16, kernel_size=5, activation=activations.relu, padding="valid")(inp)
img_1 = Convolution1D(16, kernel_size=5, activation=activations.relu, padding="valid")(img_1)
img_1 = MaxPool1D(pool_size=2)(img_1)
img_1 = Dropout(rate=0.1)(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = MaxPool1D(pool_size=2)(img_1)
img_1 = Dropout(rate=0.1)(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = MaxPool1D(pool_size=2)(img_1)
img_1 = Dropout(rate=0.1)(img_1)
img_1 = Convolution1D(256, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = Convolution1D(256, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = GlobalMaxPool1D()(img_1)
img_1 = Dropout(rate=0.2)(img_1)
dense_1 = Dense(64, activation=activations.relu, name="dense_1")(img_1)
dense_1 = Dense(64, activation=activations.relu, name="dense_2")(dense_1)
dense_1 = Dense(nclass, activation=activations.softmax, name="dense_3_mitbih")(dense_1)
model = models.Model(inputs=inp, outputs=dense_1)
opt = optimizers.Adam(0.001)
model.compile(optimizer=opt, loss=losses.sparse_categorical_crossentropy, metrics=['acc'])
model.summary()
return model
model = get_model_mitbih()
file_path = "/content/drive/My Drive/kaggle_ECG/baseline_cnn_mitbih.h5"
checkpoint = ModelCheckpoint(file_path, monitor='val_acc', verbose=1, save_best_only=True, mode='max')
early = EarlyStopping(monitor="val_acc", mode="max", patience=5, verbose=1)
redonplat = ReduceLROnPlateau(monitor="val_acc", mode="max", patience=3, verbose=2)
callbacks_list = [checkpoint, early, redonplat] # early
model.fit(X, Y, epochs=1000, verbose=2, callbacks=callbacks_list, validation_split=0.1)
model.load_weights(file_path)
pred_test = model.predict(X_test)
pred_test = np.argmax(pred_test, axis=-1)
f1 = f1_score(Y_test, pred_test, average="macro")
print("Test f1 score : %s "% f1)
acc = accuracy_score(Y_test, pred_test)
print("Test accuracy score : %s "% acc)
結果
- Test f1 score : 0.9158830356755775
- Test accuracy score : 0.9850630367257446
先ほどのMITBIHデータセットで得られたNNを利用して心筋梗塞2値分類タスクについてPTBDBデータセットで学習します。
論文では不整脈分類タスクのNNの最後の2層のみFine-tuningしてましたが、今回使うGithubのほうでは最後の2層以外の重みを固定するということはしないで、一緒に学習しなおすということをして実際最終2層より以前の重みをフリーズして学習するよりもスコアが良かったのでそちらを紹介します。
from keras import optimizers, losses, activations, models
from keras.callbacks import ModelCheckpoint, EarlyStopping, LearningRateScheduler, ReduceLROnPlateau
from keras.layers import Dense, Input, Dropout, Convolution1D, MaxPool1D, GlobalMaxPool1D, GlobalAveragePooling1D, \
concatenate
from sklearn.metrics import accuracy_score, f1_score
from sklearn.model_selection import train_test_split
df_1 = pd.read_csv("/content/drive/My Drive/kaggle_ECG/ptbdb_normal.csv", header=None)
df_2 = pd.read_csv("/content/drive/My Drive/kaggle_ECG/ptbdb_abnormal.csv", header=None)
df = pd.concat([df_1, df_2])
df_train, df_test = train_test_split(df, test_size=0.2, random_state=1337, stratify=df[187])
Y = np.array(df_train[187].values).astype(np.int8)
X = np.array(df_train[list(range(187))].values)[..., np.newaxis]
Y_test = np.array(df_test[187].values).astype(np.int8)
X_test = np.array(df_test[list(range(187))].values)[..., np.newaxis]
def get_model_ptbdb():
nclass = 1
inp = Input(shape=(187, 1))
img_1 = Convolution1D(16, kernel_size=5, activation=activations.relu, padding="valid")(inp)
img_1 = Convolution1D(16, kernel_size=5, activation=activations.relu, padding="valid")(img_1)
img_1 = MaxPool1D(pool_size=2)(img_1)
img_1 = Dropout(rate=0.1)(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = MaxPool1D(pool_size=2)(img_1)
img_1 = Dropout(rate=0.1)(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = Convolution1D(32, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = MaxPool1D(pool_size=2)(img_1)
img_1 = Dropout(rate=0.1)(img_1)
img_1 = Convolution1D(256, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = Convolution1D(256, kernel_size=3, activation=activations.relu, padding="valid")(img_1)
img_1 = GlobalMaxPool1D()(img_1)
img_1 = Dropout(rate=0.2)(img_1)
dense_1 = Dense(64, activation=activations.relu, name="dense_1")(img_1)
dense_1 = Dense(64, activation=activations.relu, name="dense_2")(dense_1)
dense_1 = Dense(nclass, activation=activations.sigmoid, name="dense_3_ptbdb")(dense_1)
model = models.Model(inputs=inp, outputs=dense_1)
opt = optimizers.Adam(0.001)
model.compile(optimizer=opt, loss=losses.binary_crossentropy, metrics=['acc'])
model.summary()
return model
model = get_model_ptbdb()
file_path = "/content/drive/My Drive/kaggle_ECG/baseline_cnn_ptbdb_transfer_fullupdate.h5"
checkpoint = ModelCheckpoint(file_path, monitor='val_acc', verbose=1, save_best_only=True, mode='max')
early = EarlyStopping(monitor="val_acc", mode="max", patience=5, verbose=1)
redonplat = ReduceLROnPlateau(monitor="val_acc", mode="max", patience=3, verbose=2)
callbacks_list = [checkpoint, early, redonplat] # early
model.load_weights("/content/drive/My Drive/kaggle_ECG/baseline_cnn_mitbih.h5", by_name=True)
model.fit(X, Y, epochs=1000, verbose=2, callbacks=callbacks_list, validation_split=0.1)
model.load_weights(file_path)
pred_test = model.predict(X_test)
pred_test = (pred_test>0.5).astype(np.int8)
f1 = f1_score(Y_test, pred_test)
print("Test f1 score : %s "% f1)
acc = accuracy_score(Y_test, pred_test)
print("Test accuracy score : %s "% acc)
結果
- Test f1 score : 0.995249406175772
- Test accuracy score : 0.9931295087598764
Preprocessingパートでみたように基線がゆれるようなデータではスケーリングがばらばらになるので論文のアルゴリズムだと十分に心拍がとれない。実際のデータにこのアルゴリズムをつかって心拍を抽出するとほんの一部分にしか推論を行えないことになる。
また、ST上昇のようなケースではST波の方がRピークよりもでかいことがあり、そのような場合にはST波の頂上をRピークと勘違いしてデータセットに入れることになるのでこれもまた論文のアルゴリズムではカバーできていない。
学習データとして心電図のII誘導しか用いていない。少ないデータで推論が行えるという利点を裏返せばこの予測器を適応できるデータは非常に限られるということだ。
またクラスの不均衡、患者のTrainとTestでのオーバーラップなどデータ分布においても問題はあって、実際の臨床心電図データに活用できるかといわれれば懐疑的である。
今回の論文では1-D CNNで心電図波形の分類を行った。転移学習の利点、学習しやすい形にする前処理の方法、心電図波形の扱い方を学んだ。比較的単純なNNモデルでも強力な予測器にすることができることも分かった。
一方好成績というのはあくまでもそのデータセットやTrain/Test分割後のバリデーションにおいてのことだけであって、Kaggle勉強会でやったように実世界の問題に基づいたバリデーションの枠組みやデータセット・データ分布の設計がなされていないと得られた予測器はいわゆる「井の中の蛙大海を知らず」ということになる。
実臨床で使える医療AIを設計するためには、実臨床データの分布となるべく似たデータセットやそれらを抽出するための前処理アルゴリズムがまず必要であるということを肝に銘じて終わる。